- 유전자 발현에 중요한 RNA-단백질간의 결합의 원리 규명 기대 -
생명 현상 조절에 필수적인 단백질의 RNA 결합자리를 RNA 염기 종류마다 특이적으로 알 수 있게 됐다. 서울대학교(총장 오세정) 자연과학대학 생명과학부 김종서 교수·김빛내리 교수 연구팀은 광활성 RNA 표지법과 화학적 RNA 분해법을 접목해, 사람 세포 내 단백질 상에서 RNA와 결합을 형성하는‘RNA 결합자리’를 RNA 염기 종류에 따라 특이적으로 정확하게 찾아낼 수 있는 질량분석 기법을 개발했다. 본 연구결과는 유전자 발현을 조절하는 RNA-단백질간 동적 상호작용을 살아있는 세포 환경에서 단일아미노산 수준의 고해상도로 분석하는 신기술로, 기존 방법으로 규명할 수 없었던 RNA-단백질 상호작용의 동적 변화와 분자적 기전 및 세포 기능 조절에 중추적인 역할을 하는 RNA-단백질 상호작용의 원리를 규명하는 데 크게 기여할 것으로 기대된다.
배종우 연구원(서울대 생명과학부)이 단독 제1저자로 참여한 이번 연구는 국제학술지‘네이처 커뮤니케이션즈 (Nature Communucations, IF=14.92) 誌에 10월 15일자 온라인판에 출간되었다 (논문명: Photoactivatable ribonucleosides mark base-specific RNA-binding sites).
세포의 정상적인 유전자 발현을 위해, 단백질은 RNA에 붙어 번역 효율, 안정성, 세포 내 위치 등을 조절한다. 이 때 RNA 결합자리는 복잡한 단백질 구조 속에서 RNA와의 결합을 매개하는 핵심적인 요소이다. 작년에 보고된 본 연구진은 기존 방법론에서는 먼저 254 nm 파장의 자외선(UVC)를 이용 해 RNA 내 유라실과 단백질 사이 교차결합을 형성한다. 이후 단백질은 효소를 이용해 펩타이드로 만들고, RNA는 불산을 이용해 유리딘으로 화학 분해한다. 결과적으로 펩타이드에 결합한 유리딘 조각의 질량을 이용해, 펩타이드 내 개별 아미노산을 RNA 결합자리로 특정할 수 있다.
그런데 기존 연구에서 사용한 UVC는, 그 유용성과 범용성에도 불구하고 복합적인 RNA-단백질 상호작용을 파악하는 데에 한계를 지닌다. 먼저 RNA-단백질 교차결합 형성 효율이 매우 낮다. 또한, 교차결합 가능한 RNA 염기 종류가 유라실 단 하나의 종류로 국한되어 있다.
연구진은 UVC 대신 광활성 RNA 표지법과 365 nm 파장의 자외선(UVA)에 의한 교차결합을 활용해 위와 같은 한계들을 극복했다. 세포 배양을 통해 세포 내 RNA를 4-티오유리딘과 6-티오구아노신 두 종류의 광활성 RNA로 표지한 후, UVA를 이용해 RNA-단백질간의 특이적 교차결합을 유도했다. 이후 단백질을 펩타이드로 가수분해하고 결합되어 있는 긴 RNA의 인산디에스터 결합을 불산 처리를 통해 화학적으로 가수 분해한다. 이 후, 질량분석을 이용해 불산에 의한 광활성 RNA 화학분해 산물의 분자량을 규명했다. 이 정보를 토대로 4-티오유리딘과 6-티오구아노신 각각 염기 종류에 해당하는 특이적 RNA 결합자리를 질량분석 기반의 단백체학 기법을 적용해 고효율로 동정해냈다. 또한, 데이터 분석 프로그램을 발전시켜 RNA 결합자리를 포함하는 펩타이드의 정확한 정량을 가능하게 했다.
새로 발견한 광활성 RNA 결합자리 뿐 아니라 기존의 UVC를 이용한 RNA 결합자리까지 망라하여, 연구진은 염기 종류 및 교차결합 방식에 따른 RNA-단백질 상호작용 양상의 차이를 면밀히 규명했다. 나아가 RNA-단백질 복합체 구조 데이터를 접목해, 질량분석으로 규명한 RNA 결합자리가 밝혀진 단백질-RNA 구조와 일치하는 염기 종류와 상호작용함을 확인했다. 또, 단백질의 구조가 불안정하여 기존의 구조 데이터만으로는 파악할 수 없는 RNA-단백질 상호작용도 RNA 결합자리를 통해 밝혀냈다.
이번에 개발한 질량분석 기법과 새로 동정한 3,000개 이상의 RNA 결합자리 정보는 RNA-단백질 상호작용에 의한 생명 현상 조절 기전의 이해를 돕는 기술 및 자원으로 높은 가치를 지닌다. 특히 광활성 RNA의 대사표지와 최근에 획기적으로 개선된 단백질 구조 예측 프로그램들을 접목하면, RNA-단백질 상호작용의 동적 변화와 그 분자적 기전을 세밀히 이해할 수 있으리라 기대된다.
연구결과
[논문명]
Photoactivatable ribonucleosides mark base-specific RNA-binding sites
Jong Woo Bae, Sangtae Kim, V. Narry Kim* and Jong-Seo Kim*
(Nature Communications, 12, Article number: 6026)
[연구 배경]
RNA-단백질 상호작용은 세포 내 다양한 생명 현상을 광범위하게 조절하는데, 이를 연구하기 위해서는 RNA결합자리를 폭넓게 밝히는 것이 매우 중요하다. 기존에 연구진은 잘량분석에 기반한 RNA 결합자리 동정기법(RBS-ID)을 가장 범용적으로 사용되는 UVC 교차결합에 대해 정립하였다. 그런데 UVC는 RNA-단백질 교차결합을 형성하는 효율이 낮았고, RNA 염기 종류가 유리딘으로 제한되어 있다는 한계를 가지고 있었다.
[연구 과정]
광활성 RNA는 염기 내 산소 원자 하나가 황 원자로 치환되어, UVC보다 파장이 비교적 긴 UVA에 의해 활성화되어 단백질과 교차결합을 형성한다. 광활성 RNA의 종류로는 4-티오유리딘과 6-티오구아노신 두 가지가 있는데, 전자의 경우 UVC에 의한 유라실 교차결합보다 그 효율이 월등히 뛰어나며, 후자를 활용하면 기존에는 보지 못했던 구아닌 염기의 RNA 결합자리를 확인할 수 있다. 연구진은 세포에 각각의 광활성 RNA를 대사표지해 RNA-단백질 사이 UVA 교차결합을 유도한 후, 펩타이드-RNA 결합체를 추출하였다. 이후 불산을 처리하여 RNA를 화학적으로 분해하고, 질량분석을 이용해 펩타이드에 결합한 광활성 RNA 조각의 분자량을 정확히 측정하였다. 연구진은 이를 정보를 토대로 각 광활성 RNA 결합자리를 구분하여 동정할 수 있는 분석 프로그램을 만들었다. 또한 각 RNA 결합 자리에 대한 정량 정보를 제공할 수 있게 프로그램을 개선했다. 이러한 실험 및 분석 파이프라인을 통칭해 pRBS-ID라 명명했다.
연구진은 기존의 UVC를 이용한 RNA 결합 자리도 개선된 프로그램으로 새로 분석해, 염기 종류 및 교차결합 방식에 따른 RNA-단백질 상호작용 양상의 차이를 도메인 및 개별 아미노산 수준에서 확인했다. 또한 기존에 알려진 RNA-단백질 복합체 구조 데이터에서도 RNA 결합자리가 실제로 각각에 해당하는 염기 종류와 상호작용한다는 점을 확인했다. 이 외에도 단백질이 불안정한 구조를 지니고 있어 기존의 구조 데이터만으로는 파악할 수 없는 RNA-단백질 상호작용도 이번에 동정한 RNA 결합 자리를 토대로 새로이 규명해냈다.
[어려웠던 점]
UVC에 의해 교차결합된 펩타이드-RNA 결합체는 실리카를 이용해 손쉽게 추출해낼 수 있다. 그런데 pRBS-ID에서 UVA 및 광활성 RNA를 사용한 경우에는 실리카로 그 결합체를 추출할 수 없었다. 연구진은 여러 시행착오 끝에 실리카 대신 올리고-d(T)와 분자량 필터를 복합적으로 활용해 펩타이드-RNA 복합체를 추출해 내는 데 성공했다.
[향후 연구계획]
pRBS-ID 및 RBS-ID를 복합적으로 활용해, RNA-단백질 상호작용 양상이 세포가 놓인 다양한 환경 및 자극 속에서 어떻게 변하는지를 탐구하고자 한다. 또한, pRBS-ID 분석 기법의 개발 과정에 착안하여 단백질과 생분자 간 다른 형태의 교차결합도 질량분석을 통해 규명해내고자 한다.
용어설명
1. 광활성 RNA (Photoactivatable ribonucleosides)
○ 염기 내 산소 원자 하나가 황 원자로 치환되어, 특정 파장의 자외선에 의해 활성화되어 단백질과 교차결합을 형성하는 RNA로서, 4-티오유리딘과 6-티오구아노신이 있으며 이는 각각 각각 유리딘 및 구아노신에 대응됨
2. RNA 결합자리 (RNA-binding site)
○ RNA 결합단백질에서 RNA를 직접 인지하여 결합하는 아미노산 자리
3 UVC
○ RNA-단백질 교차결합을 형성할 수 있는 짧은 파장(254 nm)의 자외선
4. UVA
○ 광활성 RNA-단백질 교차결합을 형성할 수 있는 비교적 긴 파장(365 nm)의 자외선
그림설명
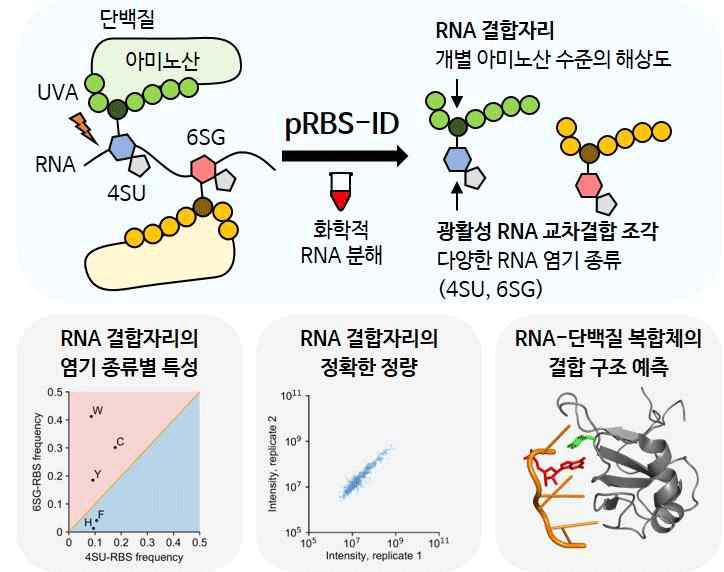
그림 1. 광활성 RNA를 이용한 pRBS-ID 연구 결과 모식도.
pRBS-ID를 위해서는 먼저 UVA를 이용해 광활성 RNA-단백질 간 공유결합을 형성한다. 그 뒤 단백질은 펩타이드로 만들고, RNA는 불산을 이용해 화학적으로 분해한다. 이후 질량분석을 수행하면 펩타이드에 교차결합한 광활성 RNA 조각의 질량으로부터 RNA 결합자리를 개별 아미노산 수준의 고해상도로 동정할 수 있다. 연구진은 pRBS-ID를 활용한 본 연구에서 RNA 결합자리의 염기 종류별 특성을 파악했고, RNA 결합자리를 정량값을 정확히 측정했으며, RNA-단백질 복합체의 구조 데이터에 기반한 RNA-단백질 상호작용 분석의 기반을 마련했다.
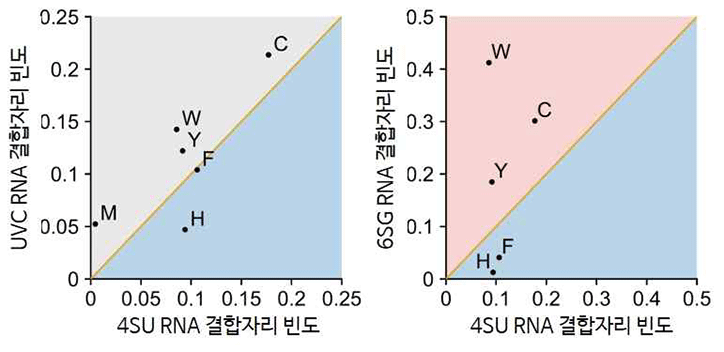
그림 2. 교차결합 방식 및 염기 종류에 따른 RNA 결합자리의 아미노산 빈도 비교.
(왼쪽) UVC에 의해 형성된 RNA 결합자리와, UVA 및 4-티오유리딘(4SU)에 의해 형성된 RNA 결합자리의 아미노산 빈도 비교. UVC의 경우 메티오닌(M)의 빈도가, 4SU의 경우 히스티딘(H)의 빈도가 특이적으로 높다. (오른쪽) UVA 및 4-티오유리딘(4SU) 혹은 6-티오구아노신(6SG)에 의해 형성된 RNA 결합자리의 아미노산 빈도 비교. 4SU의 경우 히스티딘(H)과 페닐알라닌(F)의 빈도가, 6SG의 경우 트립토판(W), 시스테인(C), 타이로신(Y)의 빈도가 특이적으로 높다.
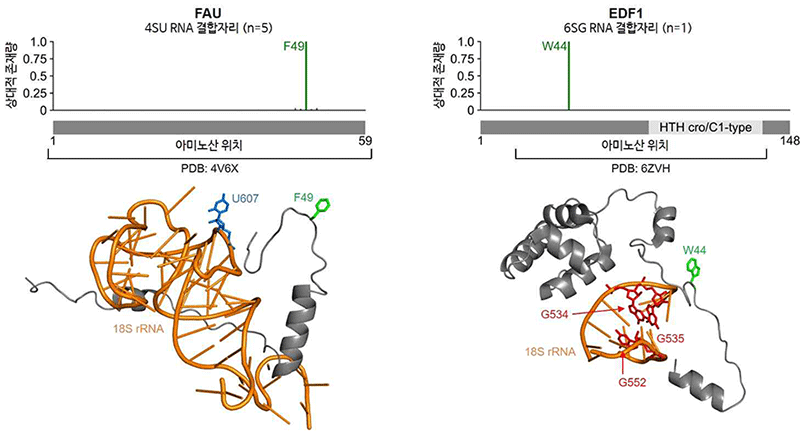
그림 3. pRBS-ID를 이용한 구조가 불안정한 단백질 부위의 RNA 결합자리 동정.
pRBS-ID 기법을 사용해 불안정성으로 인해 기존의 구조 데이터에서는 확인하기 어려웠던 RNA 결합자리를 새로 동정했다. 위쪽에는 단백질 서열상 RNA 결합자리의 위치 및 상대적 정량값이, 아래쪽에는 기존의 RNA-단백질 복합체 구조에서 RNA 결합자리 및 상호작용 가능성이 높은 RNA 염기가 표시되어 있다. (왼쪽) 리보솜의 주요 구성 단백질인 FAU의 49번 페닐알라닌(F49)와 18S rRNA의 607번 유라실(U607)이 결합 가능하다. (오른쪽) 리보솜 조절에 중요한 단백질인 EDF1의 주요 구성 단백질인 FAU의 44번 트립토판(W44)와 18S rRNA의 534/535/552번 구아닌(G534/535/552)들이 결합 가능하다.

그림 4. 연구진 사진.
왼쪽부터 김빛내리 교수(공동교신저자), 배종우 연구원(제1저자), 김종서 교수(공동교신저자). 인물 뒷 쪽 기기는 본 프로젝트에 사용한 질량분석기이다 (Orbitrap Fusion Lumos, Thermo Fisher Scientific).

